NCBI 公開收錄物種基因資訊,找到目標序列後,可透過本文列出四種方法來下載使用。
讓我們找個實際例子開始,下面是搜尋 Escherichia coli str. K-12 substr. MG1655 其中一個 assembly 的頁面
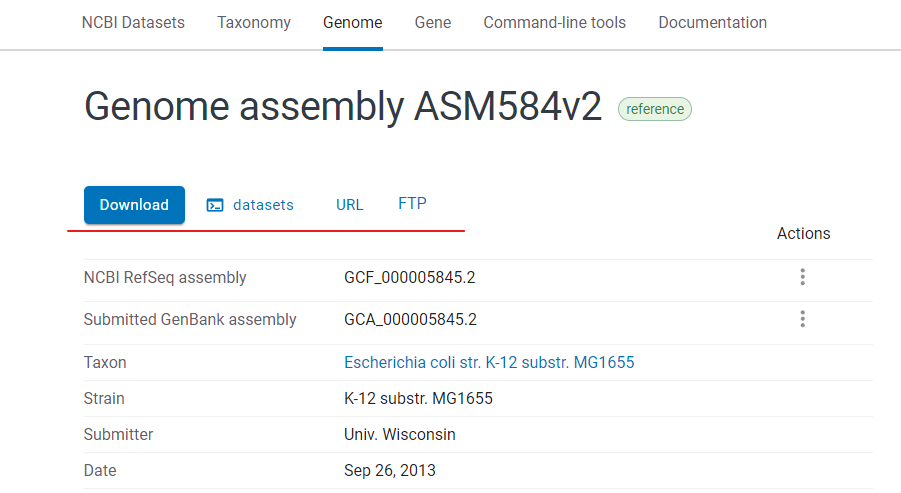
圖一: 搜尋 Escherichia coli 其中一個 assembly 的頁面截圖
紅線標示處為NCBI提供的下載管道,由左至右分別是:
- Direct Download
- Command line tools
- API URL
- FTP
Direct Download
直接下載非常直觀,選擇想要的來源(RefSeq, GenBank)和格式(FASTA,GTF…),就會獲得一個zip檔
但若序列檔案過大,或是還有二次傳輸(到server)的需求,可以考慮其他方法
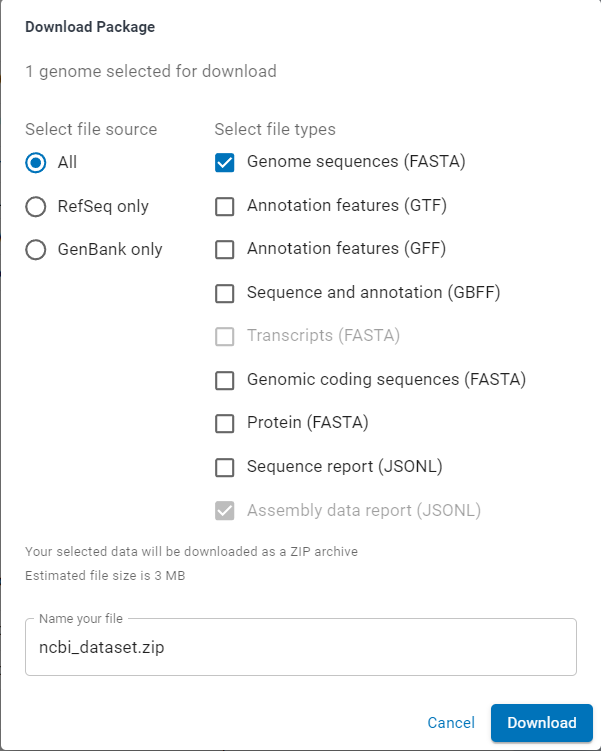
圖二: 直接下載的各種選項
NCBI 自己有個好用的工具 NCBI Datasets Command line interface (CLI) tool reference,分為協助檔案格式轉換(dataformat)和查詢下載資料集(datasets)兩大類型
注意: NCBI回傳格式為JSON,才會需要dataformat協助轉成XML, metadata等其他格式
工具安裝
工具安裝方式多元,可去NCBI官網挑選,以下使用conda安裝:
1
2
3
4
5
6
|
## create new env & activate
conda create -n ncbi_datasets
conda activate ncbi_datasets
## install via conda
conda install -c conda-forge ncbi-datasets-cli
|
完成後在cml輸入datasets 或 datafromat 測試有無 help info出現
datasets 基本功能有 summary、download、rehydrate、completion
datasets summary
利用 序列ID(genome accession)、物種學名(taxon)、基因名稱或ID(gene gene_id / symbol) 等關鍵字來查詢資料庫現有資訊
假設今天想查 TP53 在人類有什麼註解資訊:
1
2
3
4
|
datasets summary gene symbol tp53 --taxon "homo sapiens"
##如果有一份基因列表,可以用 --inputfile 提供 txt 檔一次查詢
datasets summary gene symbol --inputfile symbol.txt --taxon "homo sapiens"
|
會回傳JSON字串,可以串python用 json 來整理取出資訊:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
|
{
"reports": [
{
"gene": {
"annotations": [
{
"annotation_name": "GCF_000001405.40-RS_2023_10",
"annotation_release_date": "2023-10-02",
"assembly_accession": "GCF_000001405.40",
"assembly_name": "GRCh38.p14",
"genomic_locations": [
{
"genomic_accession_version": "NC_000017.11",
"genomic_range": {
"begin": "7668421",
"end": "7687490",
"orientation": "minus"
},
"sequence_name": "17"
}
]
},
(下略)
|
datasets download
點選圖一的 datasets 就會跳出指令,複製回去簡單上手,可再根據想要的格式增減 --include 參數
1
|
datasets download genome accession GCF_000005845.2 --include gff3,rna,cds,protein,genome,seq-report
|
最終獲得一個zip檔
API URL
點選圖一的 URL 會跳出 API URL
1
|
https://api.ncbi.nlm.nih.gov/datasets/v2alpha/genome/accession/GCF_000005845.2/download?include_annotation_type=GENOME_FASTA&include_annotation_type=GENOME_GFF&include_annotation_type=RNA_FASTA&include_annotation_type=CDS_FASTA&include_annotation_type=PROT_FASTA&include_annotation_type=SEQUENCE_REPORT&hydrated=FULLY_HYDRATED
|
可根據喜好用不同方式取得並下載
使用 curl 下載
把 API URL複製到以下curl模板裡的 ${api_url} 變數 (也記得改輸出名稱 ${output_name})
1
|
curl -X GET "${api_url}" -H "accept: application/zip" -o ${output_name}.zip
|
以圖一的例子示範:
1
|
curl -X GET "https://api.ncbi.nlm.nih.gov/datasets/v2alpha/genome/accession/GCF_000005845.2/download?include_annotation_type=GENOME_FASTA&include_annotation_type=GENOME_GFF&include_annotation_type=RNA_FASTA&include_annotation_type=CDS_FASTA&include_annotation_type=PROT_FASTA&include_annotation_type=SEQUENCE_REPORT&hydrated=FULLY_HYDRATED" -H "accept: application/zip" -o genome.zip
|
會看到下載進度:
1
2
3
|
% Total % Received % Xferd Average Speed Time Time Time Current
Dload Upload Total Spent Left Speed
100 4100k 0 4100k 0 0 1500k 0 --:--:-- 0:00:02 --:--:-- 1501k
|
完成後解壓縮取得檔案:
1
2
3
4
5
6
7
8
9
10
|
unzip genome.zip
replace README.md? [y]es, [n]o, [A]ll, [N]one, [r]ename: n
inflating: ncbi_dataset/data/assembly_data_report.jsonl
inflating: ncbi_dataset/data/GCF_000005845.2/GCF_000005845.2_ASM584v2_genomic.fna
inflating: ncbi_dataset/data/GCF_000005845.2/genomic.gff
inflating: ncbi_dataset/data/GCF_000005845.2/cds_from_genomic.fna
inflating: ncbi_dataset/data/GCF_000005845.2/protein.faa
inflating: ncbi_dataset/data/GCF_000005845.2/sequence_report.jsonl
inflating: ncbi_dataset/data/dataset_catalog.json
|
使用 python requests 下載
需要引入requests套件,把 API URL 存進 down_url 變數,以 GET 方式取得並將內容 .content 寫入 zip 檔
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
|
import requests
down_url = 'https://api.ncbi.nlm.nih.gov/datasets/v2alpha/genome/accession/GCF_000005845.2/download?include_annotation_type=GENOME_FASTA&include_annotation_type=GENOME_GFF&include_annotation_type=RNA_FASTA&include_annotation_type=CDS_FASTA&include_annotation_type=PROT_FASTA&include_annotation_type=SEQUENCE_REPORT&hydrated=FULLY_HYDRATED'
headers = {
'accept': 'application/zip',
}
response = requests.get(down_url, headers=headers)
if response.status_code == 200:
with open('genome.zip', 'wb') as fw:
fw.write(response.content)
else:
print('Fails {}'.format(response.status_code))
|
若想讓URL簡潔一些,可將 download? 問號後面的參數存成 params dict
1
2
3
4
5
6
7
8
9
10
11
12
|
down_url = 'https://api.ncbi.nlm.nih.gov/datasets/v2alpha/genome/accession/GCF_000005845.2/download'
headers = {
'accept': 'application/zip',
}
params = {
'include_annotation_type': 'GENOME_FASTA',
'include_annotation_type':'GENOME_GFF'
}
response = requests.get(down_url, headers=headers, params = params)
|
使用 REST API
直接去NCBI REST API 網頁 對照網址打上 assembly ID, include_annotation_type 等資訊,接著按照下圖順序操作:
- 點選TRY
- 確認response狀態為200
- 直接下載zip,或是複製curl指令使用
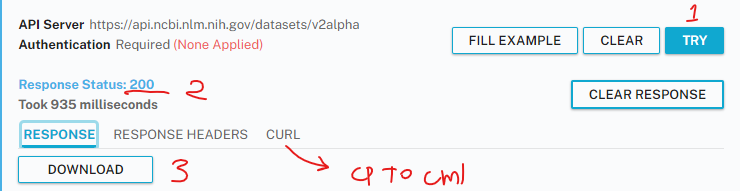
圖三: NCBI REST API 實際畫面截圖
FTP
點選圖一的FTP 就會跳轉至FTP server,可點選檔名下載,或複製檔案網址到server以 wget 取得
NCBI資料庫使用頻繁,可照個人偏好選擇方法,因最近學習 requests 和 API 相關知,有將以上code寫成 python function 來使用;而有conda建個NCBI cml 環境來玩也很方便XD



