source code 已放在我的 Github downloadDB repository: https://github.com/pxchen110/downloadDB?tab=readme-ov-file
專案動機
基因途徑(gene pathway)在生物研究扮演重要角色,如分子生物機制(molecular mechanism)、基因表現量(gene-set enrichment)的探討;研究者常參考 Kyoto Encyclopedia of Genes and Genomes (KEGG), Harmonizome 等資料庫,並具有抓取特定途徑之基因列表的需求。
但在資料取用上,對於沒有程式使用經驗(R, python)的使用者而言,常面臨以下幾個困難點:
- 資料庫僅提供 REST api 取用資料 (KEGG 和 Harmonizme)。api 處理需要串聯
requests等程式套件與網頁互動以存取資料 - 即使網站提供api資料取用,也僅提供json格式輸出(範例參考),一般文書處理程式(Microsoft Excel, Notepad++)無法直觀開啟和取用資訊
為提供更直觀方便的基因途徑探索,本專案以python為基礎處理api資訊,並以flask套件撰寫動態網頁,提供友善的html介面操作: 使用者僅需選擇資料庫來源、輸入pathway ID、貼上api網址、或上傳含有多個網址的文檔,網頁會直接回傳機因列表,並提供一鍵友善下載功能,檔案將以TXT, CSV等方便開啟的格式儲存,若提供多個列表,將會以zip壓縮檔下載給使用者。
示範操作
- 將專案 git clone 回本機,並確保本機已滿足環境需求(environment requirements):
python和conda - 進入 downloadDB 資料夾並建立 conda 環境:
conda create -f environment.yml - 執行專案前請開啟環境
conda activate flaskenv - 利用 python 開啟 flask 網頁
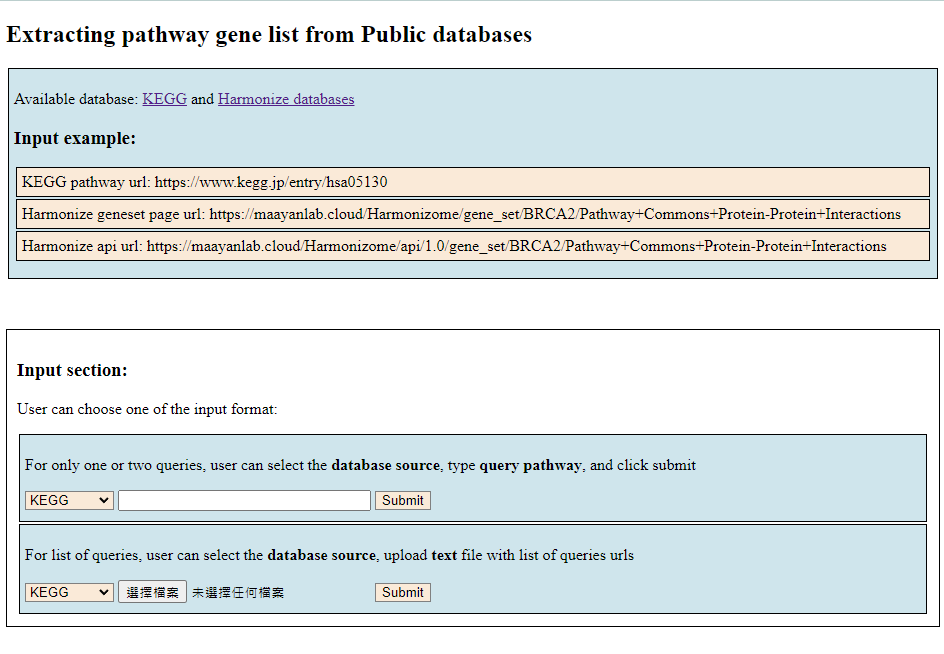
python3 app.py開啟http://127.0.0.1:5000連結後會看到以下網頁:
- 根據需求選擇其中一個Block輸入資料: !注意一次僅接受一種輸入形式!:
- 查詢一個基因途徑(single query): 選擇資料庫種類 (
KEGG或Harmonizome) 並依照範例輸入 pathway ID, api URL、點選Submit送出 - 查詢多個基因途徑(multiple query):準備一份紀錄api URL 的 TXT 檔,選擇資料庫種類 (
KEGG或Harmonizome)並上傳檔案、點選Submit送出
- 查詢一個基因途徑(single query): 選擇資料庫種類 (
- 若目標api或基因途徑存在,將會返回以下網頁內容:
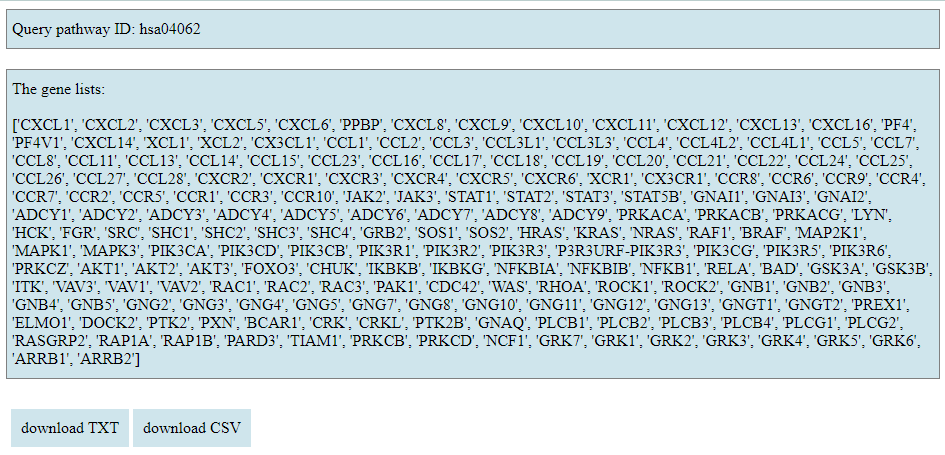
- 點選 Download TXT 或 Download CSV 下載不同格式的基因列表;若提供多個基因途徑,點選Download ZIP 下載zip壓縮檔,解壓縮後每個基因列表產生一份TXT檔
未來改善
- 由於目前python程式寫法為: 取得資料後會將基因列表儲存成txt和csv檔(專案路徑下名為
query_output和query_input的資料夾),並以檔名作為變數傳遞至下個函數;若使用者點選 download 按鈕並將檔案下載至本機,此時電腦會有兩份相同的檔案,希望能增加自動清除檔案功能 - 如何跳轉回最初頁面
- 嘗試連結後端資料庫儲存的函數功能